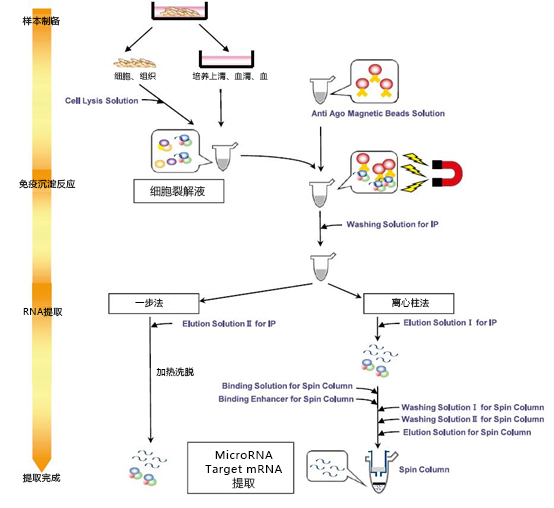
磁珠型microRNA分离试剂盒
FUJIFILM Wako提供能使从Ago 免疫沉淀到RNA 提取等一系列实验操作简化的“microRNA Isolation Kit”系列。使用本系列试剂盒能简便地从人和小鼠的细胞或组织中提取Ago蛋白结合的microRNA以及靶mRNA,还能从培养上清、血清、血浆中提取游离Ago蛋白结合的microRNA。所提取到的RNA可以应用于qPCR、微阵列检测、NGS等下游实验。本系列包括采用磁珠的MagCapture™ microRNA Isolation Kit,以及采用了硅胶珠法的microRNA Isolation Kit。

MagCapture™ microRNA Isolation Kit(磁珠法)系列产品包括含抗人Ago2抗体(4G8)、抗小鼠Ago2抗体(2D4)、抗Ago1-4抗体(1G3)固相化磁珠这三种试剂盒,以及第四种——能结合任意抗体的Protein G磁珠。上述试剂盒全部采用磁珠法,可进行简易的免疫沉淀操作并获得良好的重复性。免疫沉淀后提取的RNA可进一步使用离心柱纯化,获得高纯度的RNA;若使用含有抗Ago抗体固定化磁珠的3种试剂盒,也可使用一步法更加简便地提取RNA(备注:RNA与磁珠不分离)。
◆试剂盒组成(例:Human Ago2, 10次用)
1. Anti-Human Ago2 Magnetic Beads Solution:600 μL×1瓶
2. Cell Lysis Solution:20 mL×1瓶
3. Washing Solution for IP:40 mL×1瓶
4. Elution Solution I for IP:500 μL×1瓶
5. Elution Solution II for IP:500 μL×1瓶
6. Binding Solution for Spin Column:2 mL×1瓶
7. Binding Enhancer for Spin Column:100 μL×1瓶
8. Washing Solution I for Spin Column:3 mL×1瓶
9. Washing Solution II for Spin Column:4 mL×1瓶
10. Elution Solution for Spin Column:1 mL×1瓶
11. Spin Column/Collection Tube:10瓶
◆操作示意图
◆MagCapture™ microRNA Isolation Kit,Human Ago2 使用例子(人)
提取人细胞株中的microRNA
通过变性聚丙烯酰胺凝胶电泳(银染),检测使用本品(MagCapture™ microRNA Isolation Kit,Human Ago2)从人K562细胞(1×107 cells)中提取的microRNA(图1)。结果显示,本系列试剂盒可提取得到高纯度的microRNA。通过qPCR对miR-92a进行定量,比较使用本品和硅胶珠型产品(microRNA Isolation Kit,Human Ago2)提取的microRNA量(图2)。本系列试剂盒的离心柱法具有几乎与硅胶珠法相同的microRNA提取能力,而且一步法的microRNA提取能力比硅胶珠法更好。
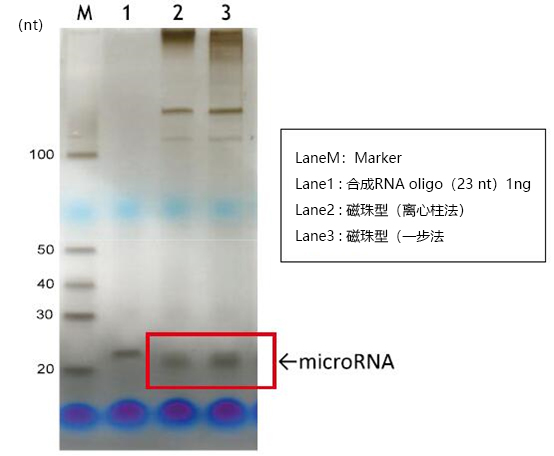
图1. 对从人K562细胞中提取的microRNA的进行银染检测
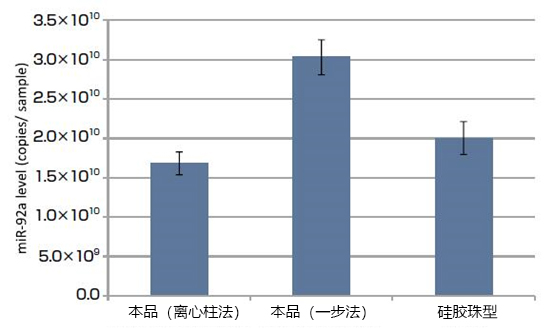
图2. 比较从人K562细胞中提取的microRNA量
提取人血浆中的游离Ago2蛋白结合microRNA
以qPCR对miR-92a进行定量,比较使用磁珠型MagCapture™ microRNA Isolation Kit,Human Ago2和硅胶珠型产品从200μL健康人混合血浆中提取的microRNA量。结果发现,本试剂盒中的离心柱法具有几乎与硅胶珠型产品相同的microRNA提取能力,而且一步法的microRNA提取量比硅胶珠型高(图3)。
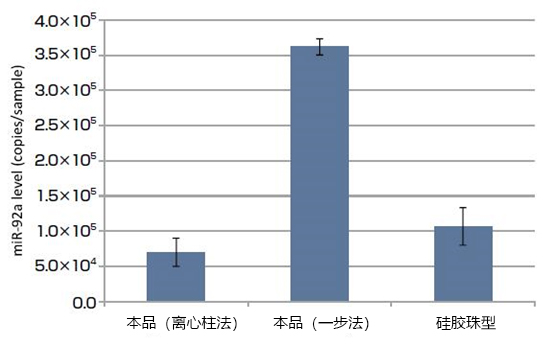
图3. 比较从人血浆中提取的microRNA量
提取microRNA的靶标mRNA
向miR-122低表达的肝癌细胞株HepG2细胞(5×105 cells)导入人工合成的miR-122或调控的荧光素酶siRNA(Luc siRNA),提取了分别用磁珠型MagCapture™ microRNA Isolation Kit,Human Ago2和硅胶珠型产品制备的Ago2 IP RNA,以及用ISOGEN® 制备的total RNA。运用qPCR法比较各种RNA中含有的miR-122量(图4)与其靶标mRNA(Aldo A mRNA是miR-122靶标之一,以GAPDH内参作校正)量(图5)。与硅胶珠型产品一样,本品提取的RNA中富集了miR-122和Aldo A mRNA,因此,可以利用本品提取microRNA的靶标mRNA。另一方面,用ISOGEN® 制备的total RNA中的Aldo A mRNA会随着miR-122的增加而分解并减少。
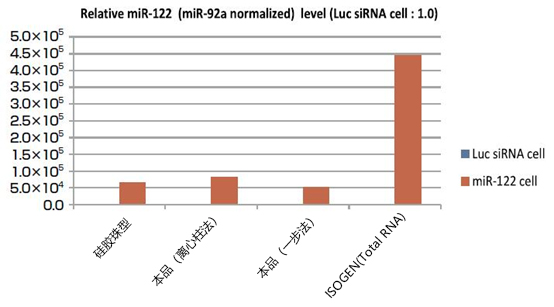
图4. miR-122提取量的比较
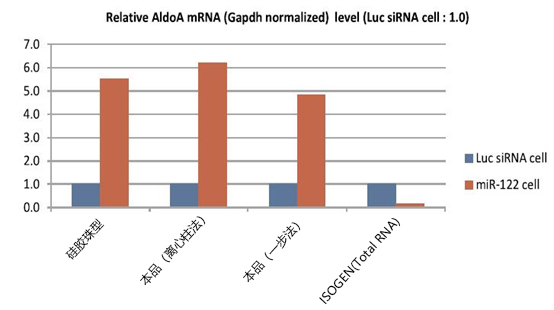
图5. 靶标mRNA提取量的比较
◆MagCapture™ microRNA Isolation Kit, Mouse Ago2 使用例子(小鼠)
从小鼠细胞株中提取microRNA
通过变性聚丙烯酰胺凝胶电泳(银染),检测用本试剂盒(MagCapture™ microRNA Isolation Kit,Mouse Ago2)从小鼠P388D1细胞(2×107 cells)中提取的microRNA(图1)。结果显示,本品能提取出高纯度的microRNA。另外,以qPCR法对miR-92a进行定量,比较使用硅胶珠型产品(microRNA Isolation Kit,Mouse Ago2)提取到的microRNA量(图2)。结果发现,本品的离心柱法具有与硅胶珠型产品几乎同样的microRNA提取能力,而一步法的microRNA提取能力比硅胶珠型产品更好。
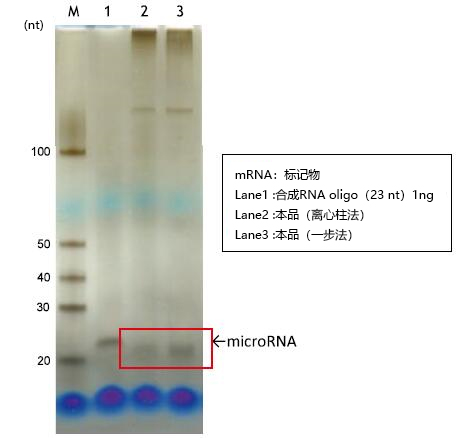
图1. 对从小鼠P388D1细胞中提取的microRNA的进行银染检测
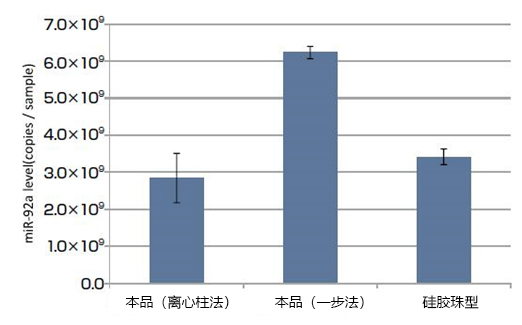
图2. 比较从小鼠P388D1细胞中提取的microRNA量
从大鼠血浆中提取游离Ago2蛋白结合microRNA
通过qPCR对miR-92a进行定量,比较使用本品以及硅胶珠型产品从200μL大鼠血浆中提取到的microRNA量。本品的离心柱法具有与硅胶珠型产品几乎同样的microRNA提取能力,而一步法的microRNA提取能力比硅胶珠型产品更好(图3)。
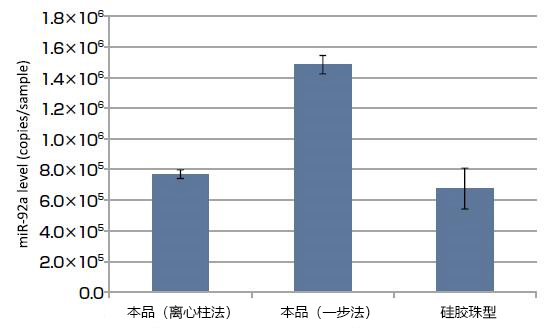
图3. 比较从大鼠血浆中提取的microRNA量
◆MagCapture™ microRNA Isolation Kit, Ago 1–4 使用例子(人)
提取Ago1–4结合microRNA
用各种Ago1、Ago2、Ago3、Ago4特异性抗体进行微阵列检测分析免疫沉淀后HeLa中的各个RNA组分,结果显示,Ago2中捕获了大量miR-22和miR-16,而Ago1、Ago3、Ago4中则捕获了大量的miR-1260b和miR-4286(表1)。
|
信号强度
|
信号比
|
|
名称
|
Ago1 IP
|
Ago2 IP
|
Ago3 IP
|
Ago4 IP
|
Ago2/Ago1
|
Ago2/Ago3
|
Ago2/Ago4
|
|
hsa-miR-22
|
3521.4
|
21696.5
|
3771.2
|
292.4
|
6.16
|
5.75
|
74.2
|
|
hsa-miR-16
|
6886.3
|
28527.2
|
5013.4
|
582.5
|
4.14
|
5.69
|
48.97
|
|
hsa-miR-1260b
|
2060.3
|
540.9
|
1270.6
|
1977.9
|
0.26
|
0.43
|
0.27
|
|
hsa-miR-4286
|
678.8
|
202.4
|
2019.2
|
471.2
|
0.3
|
0.1
|
0.43
|
表1. 微阵列检测分析结果
上述分析结果验证了我们可以利用MagCapture™ microRNA Isolation Kit, Ago1-4,从Ago1、Ago3、Ago4中提取硅胶珠型产品难以提取的大量microRNA。
使用本品和硅胶珠型产品,提取HeLa细胞(1×107 cells)中的microRNA,以miR-92a作为内部标准,通过qPCR检测miR-22、miR-1、miR-1260b、miR-4286的提取量。结果发现,Ago1、Ago3、Ago4中miR-1260b、miR-4286的提取量比硅胶珠型产品更高(图1)。因此,本品不仅能提取Ago2中的microRNA,还能从Ago1、Ago3、Ago4中提取硅胶珠型产品难以提取的大量microRNA。
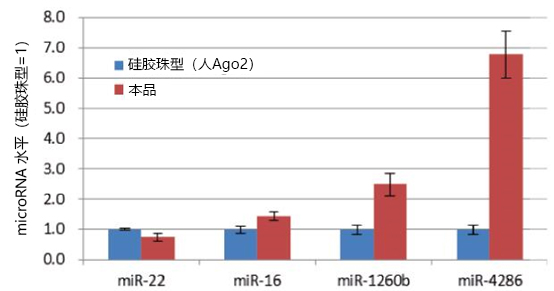
图1. 比较从HeLa细胞中提取的各种microRNA量
提取人和小鼠细胞株中的microRNA
以qPCR对miR-92a进行定量,比较用本品和硅胶珠型产品(microRNA Isolation Kit, Human Ago2或Mouse Ago2)从人K562细胞(1×107 cells)及小鼠P388D1细胞(2×107 cells)中提取的microRNA量。结果显示,本品可提取和硅胶珠型产品相同甚至更多的microRNA(图2,3)。
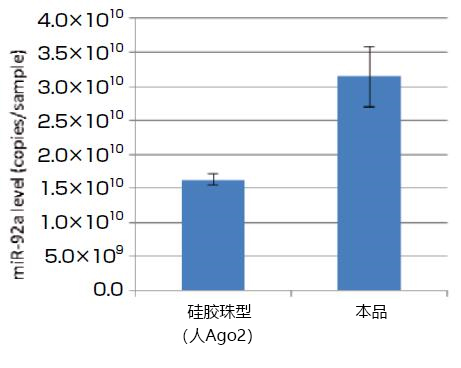
图2. 比较从人K562细胞中提取的microRNA量
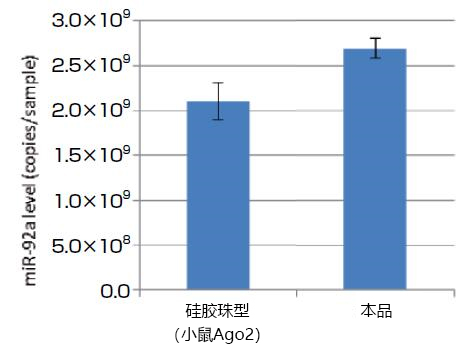
图3. 比较从小鼠P388D1细胞中提取的microRNA量
提取人和大鼠血浆中的游离Ago结合microRNA
以qPCR对miR-92a进行定量,比较用本品和第一代产品(microRNA Isolation Kit, Human Ago2或Mouse Ago2)从200μL人以及大鼠血浆中提取的microRNA量。结果显示,本品可提取和硅胶珠型产品相同甚至更多的microRNA(图4,5)。
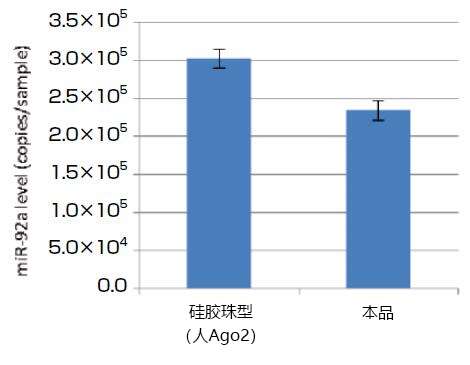
图4. 比较从人血浆中提取的microRNA量
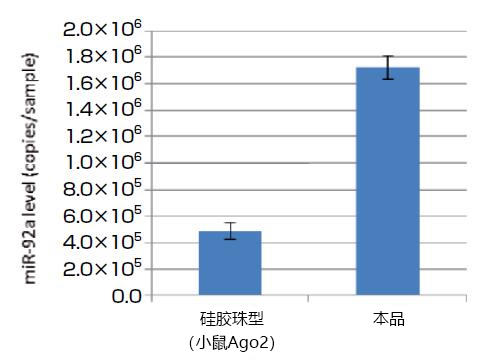
图5. 比较从大鼠血浆中提取的microRNA量
提取各种动物细胞株中的microRNA
以qPCR对miR-92a进行定量,检测用MagCapture™ microRNA Isolation Kit, Human Ago2、Mouse Ago2、Ago1-4从2×106 cells的K562细胞(人)、COS-7细胞(猴)、MDCK细胞(狗)、P388D1细胞(小鼠)、SCC-131细胞(大鼠)中提取的免疫沉淀组分中的microRNA量,和用ISOGEN® 从各种细胞中提取的total RNA组分中的microRNA量。通过比较各种免疫沉淀所得到的RNA组分中的miR-92a量和total RNA组分中的miR-92a量的相对值,表明本品适合提取上述全部细胞中的microRNA(图6)。
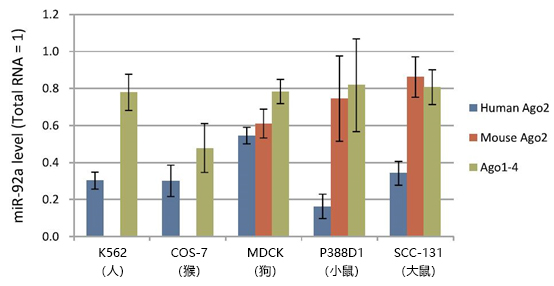
图6. 比较从各种动物细胞中提取的microRNA量
◆MagCapture™ microRNA Isolation Kit, Protein G使用例子
提取人和小鼠细胞株中的microRNA(与硅胶珠型产品的比较)
以qPCR法对miR-92a进行定量,比较用本品(MagCapture™ microRNA Isolation Kit, Protein G)和抗Ago1抗体(2A7)、抗人Ago2抗体(4G8)、抗人Ago3抗体(1C12)从人K562细胞(1×107 cells)提取的microRNA量,和用硅胶珠型产品(microRNA Isolation Kit, Human/Mouse Ago1、Human Ago2、Human Ago3)提取的micro RNA量(图1,2,3,)。另外,以qPCR对miR-92a进行定量,比较用本品和抗小鼠Ago2抗体(2D4)从小鼠P388D1细胞(2×107 cells)中提取的microRNA量,和用硅胶珠型产品(microRNA Isolation Kit, Mouse Ago2)提取的microRNA量(图4)。结果发现,本品与任意抗Ago抗体结合使用,能比硅胶珠型产品提取到更多的microRNA。
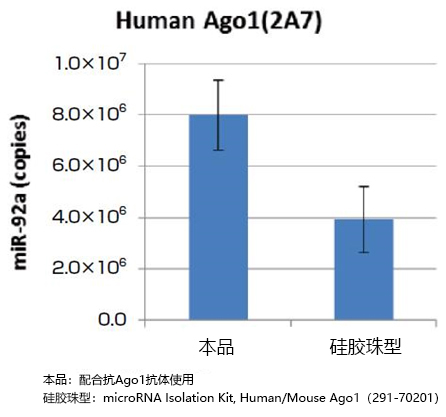
图1. 比较结合了抗Ago1抗体时的microRNA提取量
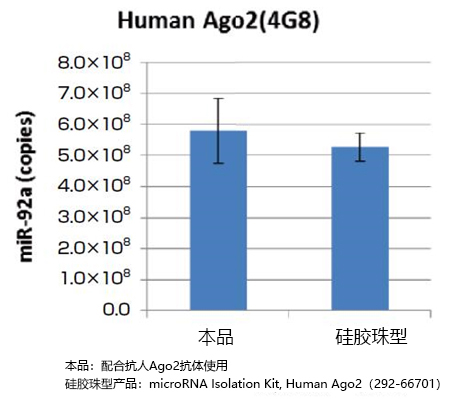
图2. 比较固定抗人Ago2抗体时的microRNA提取量
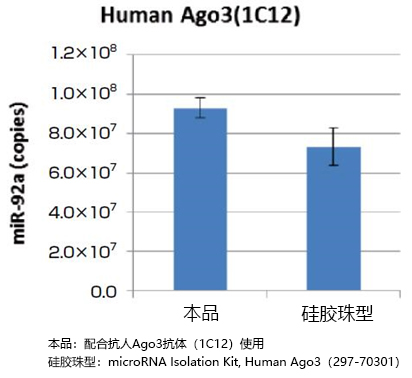
图3. 比较固定抗人Ago3抗体时的microRNA提取量
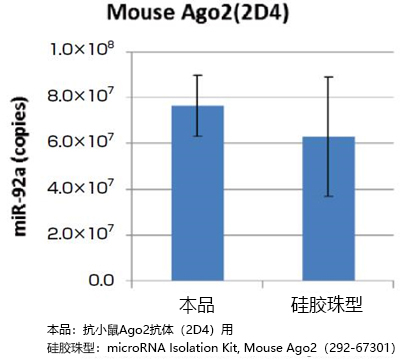
图4. 比较固定抗小鼠Ago2抗体时microRNA的提取量
提取人与小鼠细胞株中各种Ago蛋白结合microRNA
通过变性聚丙烯酰胺凝胶电泳和qPCR,对用本品和抗Ago1抗体(2A7)、抗人Ago2抗体(4G8)、抗人Ago3抗体(1C12)、抗Ago4抗体(2G7)从人K562细胞(1×107 cells)中提取的miR-92a表达量进行检测(图5)。另外,通过变性聚丙烯酰胺凝胶电泳和qPCR(miR-92a量),检测用本品和抗Ago1抗体(2A7)、抗小鼠Ago2抗体(2D4)、抗小鼠Ago3抗体(S09)、抗Ago4抗体(2G7)从小鼠P388D1细胞(2×107 cells)中提取的microRNA量(图6)。结果表明,本品与任意的抗Ago抗体组合使用,能提取与各种Ago结合的microRNA。
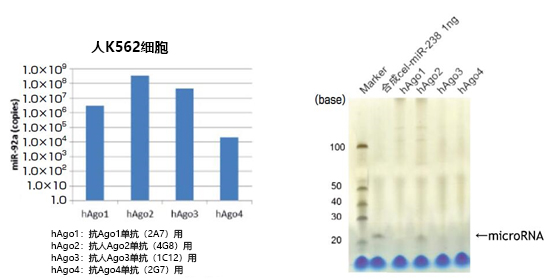
图5. 从人K562细胞中提取的microRNA量的比较与银染检测
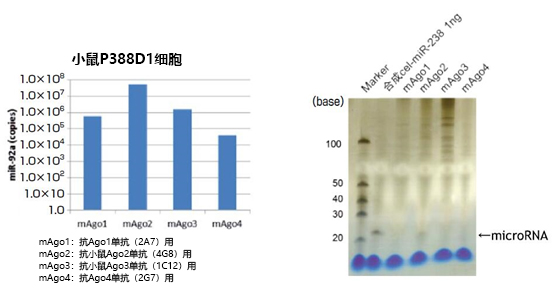
图6. 从小鼠P388D1细胞中提取的microRNA量的比较与银染检测
◆产品列表
|
产品编号
|
产品名称
|
使用
抗体
克隆号
|
对应
物种
|
对应样本
|
提取RNA
|
IP B/F
分离法
|
RNA
提取法
|
|
295-74001
|
MagCapture™ microRNA Isolation Kit, Human Ago2
MagCapture™ microRNA
分离试剂盒,人Ago2
|
4G8
|
人、猴、狗
|
细胞/
组织
血清/
血浆
|
microRNA
靶标mRNA
|
磁力
分离
|
离心
柱法
一步法
|
|
297-74201
|
MagCapture™ microRNA Isolation Kit,Mouse Ago2
MagCapture™ microRNA
分离试剂盒,小鼠Ago2
|
2D4
|
小鼠、大鼠、狗
|
|
293-74801
|
MagCapture™ microRNA Isolation Kit,Ago 1-4
MagCapture™ microRNA
分离试剂盒,Ago1-4
|
1G3
|
人、猴、狗、小鼠、大鼠
|
microRNA
|
|
299-74401
|
MagCapture™ microRNA Isolation Kit,Protein G
MagCapture™ microRNA
分离试剂盒,蛋白G
|
随意
选择
|
根据使用的抗体而定
|
细胞/
组织
|
根据使用的抗体而定
|
一步法
|
 已登记MF
已登记MF



































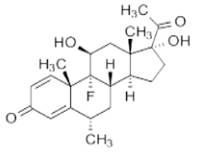

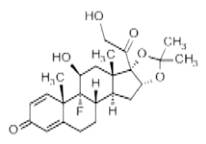
 腔肠素h
腔肠素h



























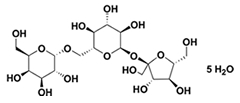 ◆D(+)-五水棉子糖
◆D(+)-五水棉子糖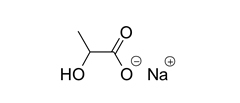 ◆
◆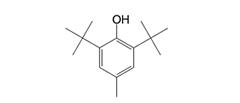 ◆
◆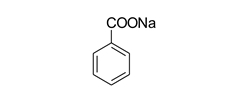 ◆
◆ ◆
◆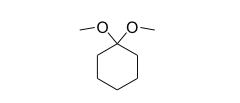 ◆
◆
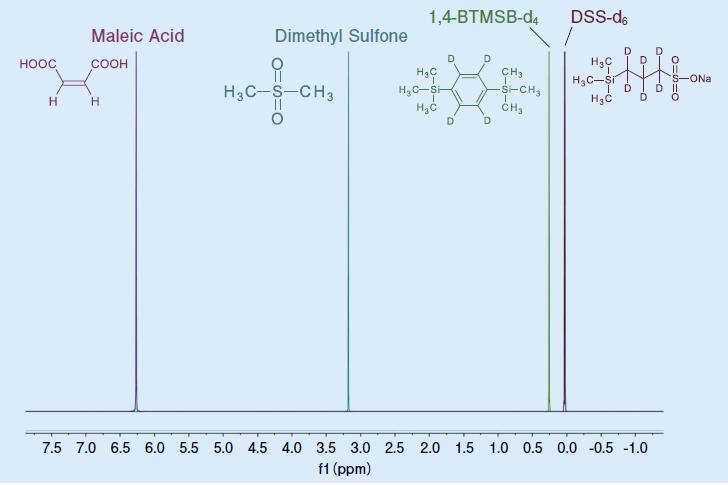
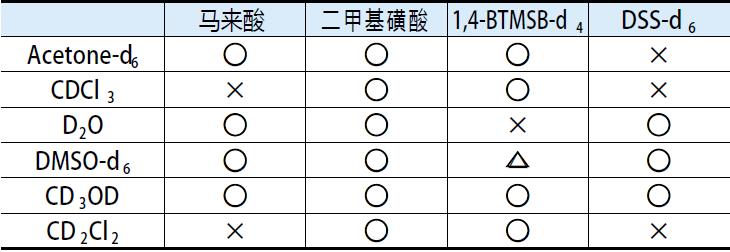
 定量NMR用内标准溶液
定量NMR用内标准溶液